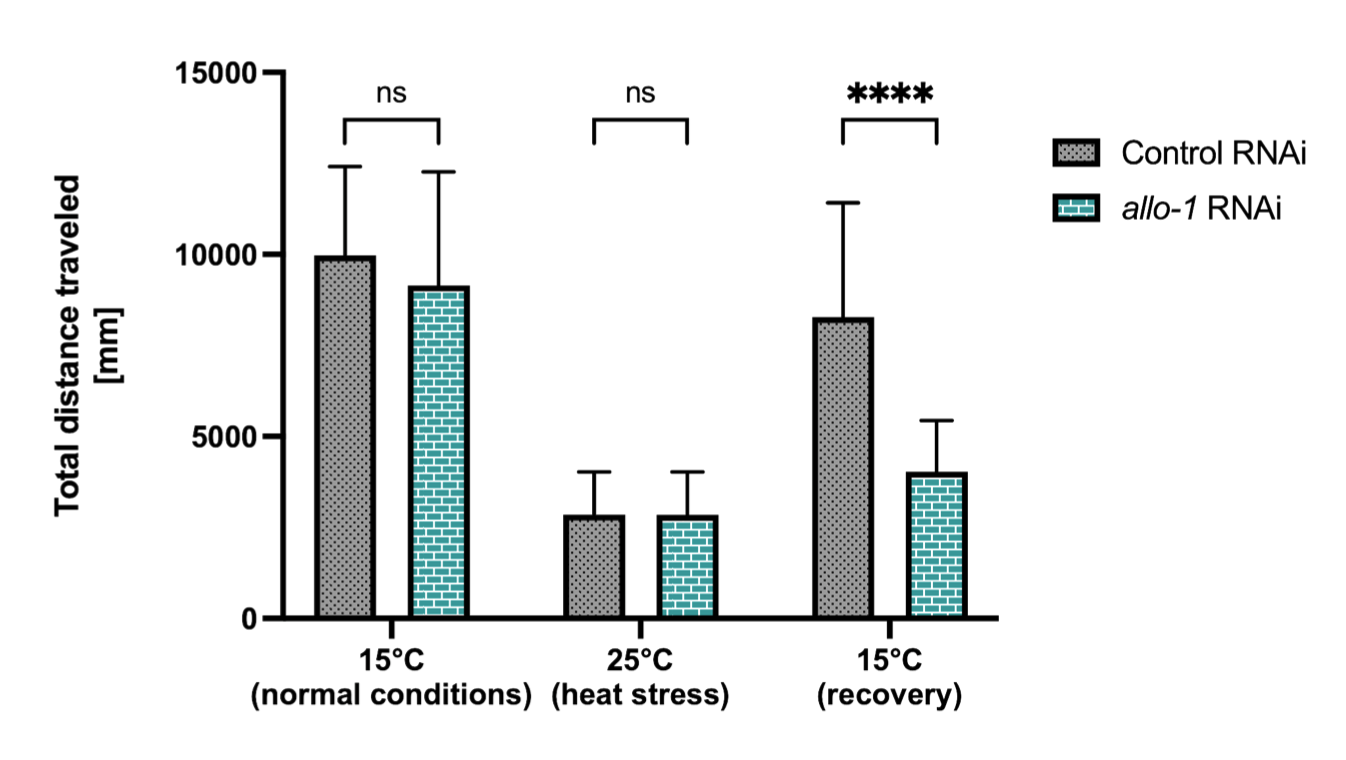
The ubiquitin-conjugating enzyme UBE2K determines neurogenic potential through histone H3 in human embryonic stem cells
Fatima, A., Irmak, D., Noormohammadi, A., Rinschen, M.M., Das, A., Leidecker, O., Schindler, C., Sánchez-Gaya, V., Wagle, P., Pokrzywa, W., Hoppe, T., Rada-Iglesias, A., & Vilchez, D.
Communications Biology
doi: 10.1038/s42003-020-0984-3
Ubiquitin signaling regulates RNA biogenesis, processing, and metabolism
Thapa, P., Shanmugam, N., & Pokrzywa, W.*
BioEssays
doi: 10.1002/bies.201900171
CHIP ubiquitylates NOXA and induces its lysosomal degradation in response to DNA damage
Albert, M.-C., Brinkmann, K., Pokrzywa, W., Günther, S. D., Krönke, M., Hoppe, T., & Kashkar, H.
Cell Death & Disease
doi: 10.1038/s41419-020-02923-x
Bioshell 3.0: Library for processing structural biology data
Macnar, J.M., Szulc, N.A., Kryś, J.D., Badaczewska-Dawid, A.E., & Gront, D.
Biomolecules
doi: 10.3390/biom10030461
Pathogenic variants in the myosin chaperone UNC-45B cause progressive myopathy with eccentric cores
Donkervoort, S., Kutzner, C. E., Hu, Y., Lornage, X., Rendu, J., Stojkovic, T., Baets, J., Neuhaus, S.B., Tanboon, J., Maroofian, R., Bolduc, V., Mroczek, M., Conijn, S., Kuntz, N. L., Töpf, A., Monges, S., Lubieniecki, F., McCarty, R. M., Chao, K. R., Governali, S., Böhm, J., Boonyapisit, K., Malfatti, E., Sangruchi, T., Horkayne-Szakaly, I., Hedberg-Oldfors, C., Efthymiou, S., Noguchi, S., Djeddi, S., Iida, A., di Rosa, G., Fiorillo, C., Salpietro, V., Darin, N., Faure, J., Houlden, H., Oldfors, A., Nishino, I., de Ridder, W., Straub, V., Pokrzywa, W., Laporte, J., Foley, R., Romero, N.B., Ottenheijm, C., Hoppe, T., & Bönnemann, C.G.
The American Journal of Human Genetics
doi: 10.1016/j.ajhg.2020.11.002
Ubiquitylation pathways in insulin signaling and organismal homeostasis
Balaji, V.†, Pokrzywa, W.†, & Hoppe, T.
BioEssays
doi: 10.1002/bies.201700223
The ubiquitin ligase UBR5 suppresses proteostasis collapse in pluripotent stem cells from Huntington’s disease patients
Koyuncu, S., Saez, I., Lee, H. J., Gutierrez-Garcia, R., Pokrzywa, W., Fatima, A., Hoppe, T., & Vilchez, D.
Nature Communications
doi: 10.1038/s41467-018-05320-3
Chaperone-directed ubiquitylation maintains proteostasis at the expense of longevity
Pokrzywa, W., Lorenz, R., & Hoppe, T.
Worm
doi: 10.1080/21624054.2017.137140
CHIPped balance of proteostasis and longevity
Pokrzywa, W., & Hoppe, T.
Oncotarget
doi: 10.18632/oncotarget.22101
Repair or destruction - an intimate liaison between ubiquitin ligases and molecular chaperones in proteostasis
Kevei, É.†, Pokrzywa, W.†, & Hoppe, T.
FEBS Letters
doi: 10.1002/1873-3468.12750
---
pogrubienie - pracownik Laboratorium Metabolizmu Białek
* autor korespondencyjny
† równy wkład